NMR化学位移、考虑自旋-自旋耦合的NMR化学位移
软件版本为AMS2019.3及之前的版本,AMS2020以后的版本请参考链接:考虑自旋-自旋耦合的1H-NMR化学位移的计算
使用ADF的图形界面,可以直接对比计算结果与实验结果:
计算前,请务必参考:
本教程使用AMS2019.301完成
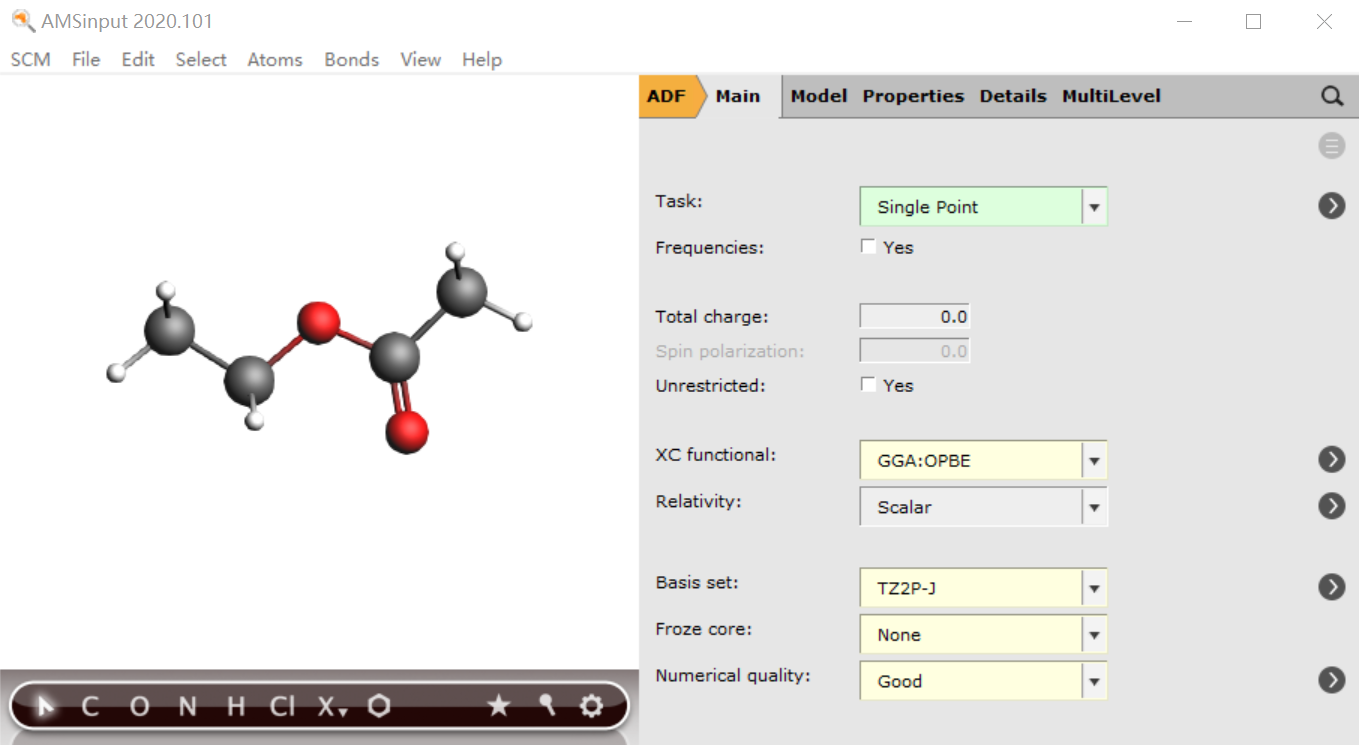
第一步:结构优化
参考:优化分子的几何结构,NMR相关计算,可以使用高精度优化,例如:
- Hybrid:PBE0
- Basis: TZP
- Frozen core: None
- Numerical Quality: Good
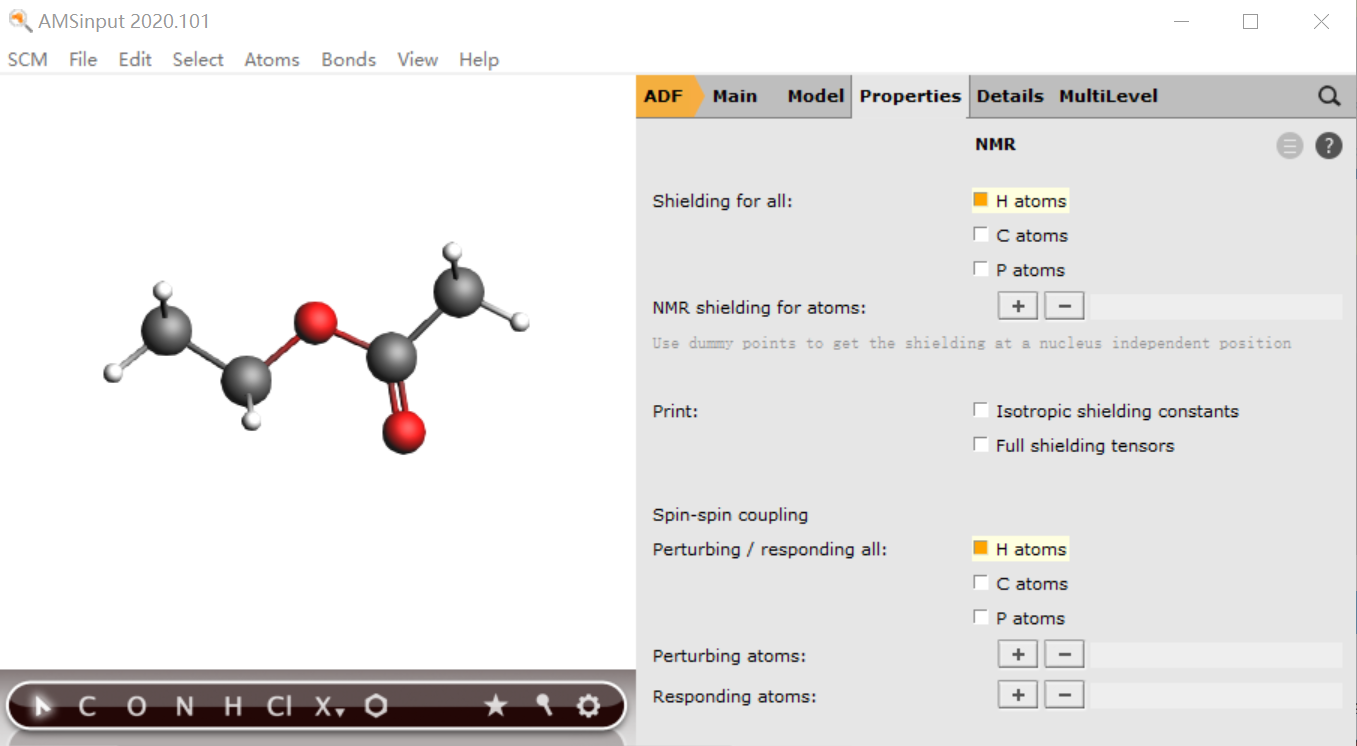
第二步:NMR计算
注意:
- 如果只需要计算其中一部分原子的NMR,可以在窗口左边选中这些原子,然后点击右方窗口NMR shielding for atoms后面的➕;
- 如果只考虑部分原子对某些原子的自旋-自旋耦合,可以分别选中这些原子,在下方的Perturbing atoms和Responding atoms后面的➕,Perturbing atoms对Responding atoms进行微扰,用户也可以不勾选上面的元素框,改为一一选择设置
- 如果不考虑自旋-自旋耦合,则不设置上图中Spin-Spin Coupling即可。
- 基组中,J-TZ2P或J-QZ4P是专用于NMR计算的基组
保存任务,并运行。
第三步:结果查看
NMR化学位移、自旋-自旋耦合常数
SCM - Output - Other Properties - Spin-Spin Coupling
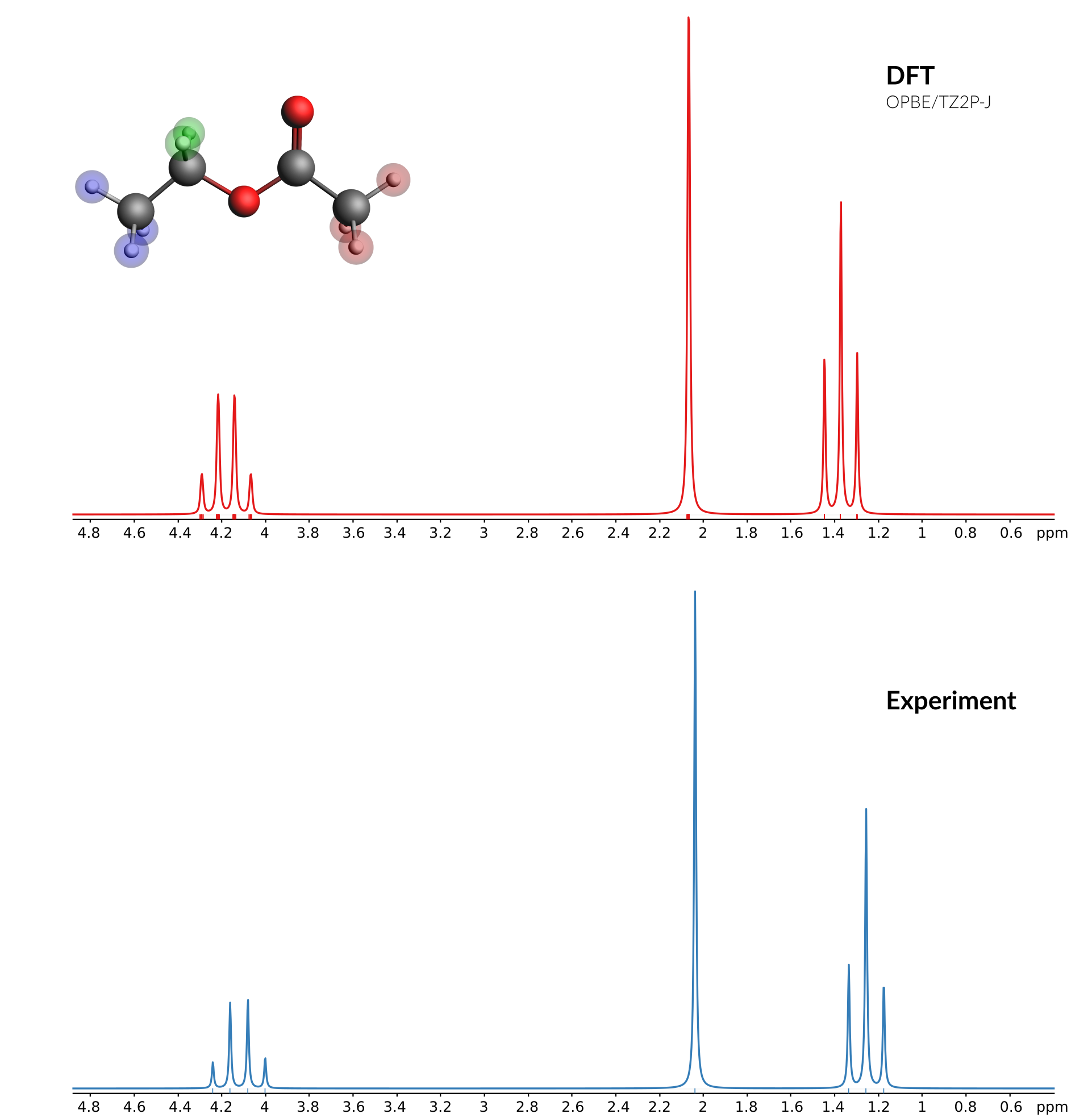
查看$^1$H-NMR谱
ADF LOGO > Spectra:
这是没有自旋-自旋耦合的结果。每个峰都对应着某个原子,点击这些原子,对应的峰也被选中了。对这个分子而言,中间的两个H原子是等价的,因此可以进行平均:
选中左边窗口的这两个H原子,按Ctrl G(或者点击菜单Regions → New Region From Selected Atoms)这样,谱图就对这两个H原子进行了平均。类似地,可以分别对两个甲基上的三个H原子进行平均。从而得到只有三个峰的谱图。
查看加入自旋-自旋耦合的$^1$H-NMR谱
与实验对比
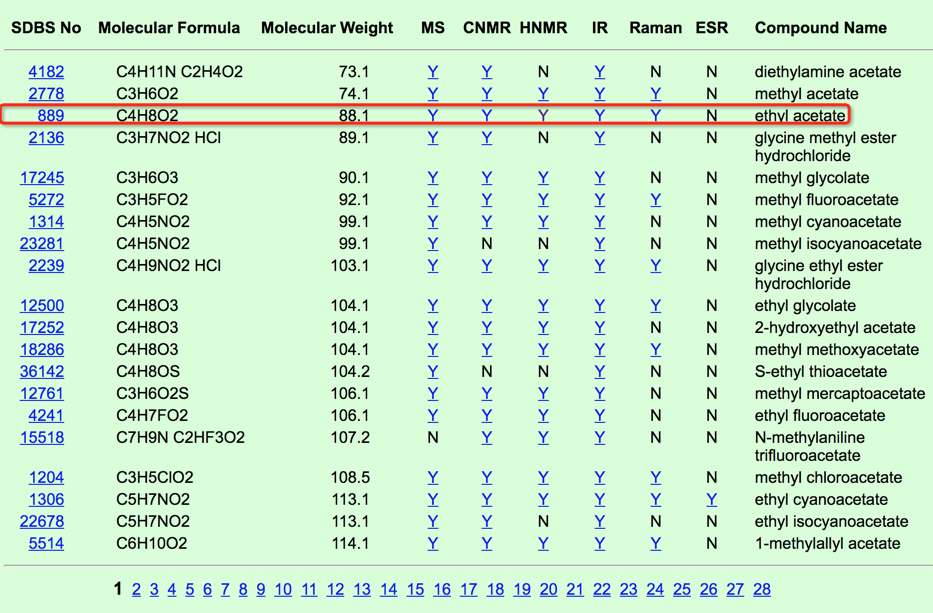
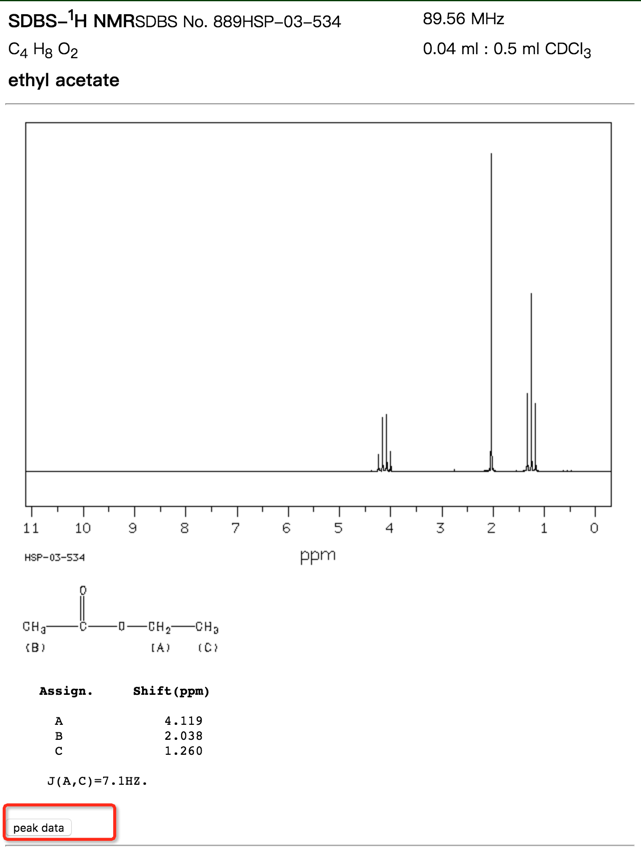
不要关闭上面的谱图窗口。在http://sdbs.db.aist.go.jp/sdbs/cgi-bin/direct_frame_top.cgi(点击窗口下方的“I agree the disclaimer and use SDBS)”按钮才能搜索。搜索“ethyl acetate”,
选择ethyl acetate的HNMR数据:
点击窗口左下角的“peak data”,复制这些数据:
379.88 4.242 52 372.75 4.163 171 365.56 4.082 180 358.50 4.003 62 182.56 2.039 1000 119.69 1.337 245 112.63 1.258 560 105.38 1.177 214
回到ADF的谱图窗口,直接ctrl v粘贴这些数据,会弹出提示,问图谱的x值是第几列数据,显然是上面的第2列,图谱的y显然是上面的第3列数据。所以在对话框分别输入2和3即可看到是实验谱和计算谱在同一个窗口了。
还应该修改Coupling后面的频率(默认值为200MHz),本例中,实验上,是用89.56Hz的信号去检测的,因此可以修改为89.56: