基于 GCMC 预测吸附等温线:CO$_2$ 在 IRMOF-1 中的吸附
关键词:MOF材料;IRMOF-1;GCMC;巨正则系综蒙特卡洛;ReaxFF
软件版本要求:AMS2023 及以上。
概述
吸附等温线表示“主体材料”在给定温度和压力下,摄入给定数量“客体分子”的性质。吸附等温线是负载与压强的函数,负载通常表示为每千克主体材料吸附多少摩尔客体分子,单位为 mol/kg)。在本教程中,我们将以通用性较好的 MOF 材料 IRMOF-1 对 CO$_2$ 的负载为例,演示如何使用巨正则系综蒙特卡洛模拟生成吸附等温线。
实际上可以使用各种力场、机器学习势、DFTB(DFTB中D3(BJ)色散修正对此应该会有较好效果)、MOAPC,甚至基于 BAND 的 DFT,使用过程基本上没有大的区别,不过本例中以 ReaxFF/CHOZn.ff 为例进行演示。CHOZn.ff 力场在 AMS2023 中尚未加入,用户可自行下载解压使用(AMS2024 版中将加入该力场)。
建模
CO$_2$
在 ReaxFF 中创建并优化 CO$_2$ 如下所示:
File → Save,保存名为例如 CO2_ref_ReaxFF,并运行。运行结束后,SCM → logfile 在尾部可以看到总能量E$_{CO2}$ = -0.6434 Hartree = -1689.155kJ/mol:
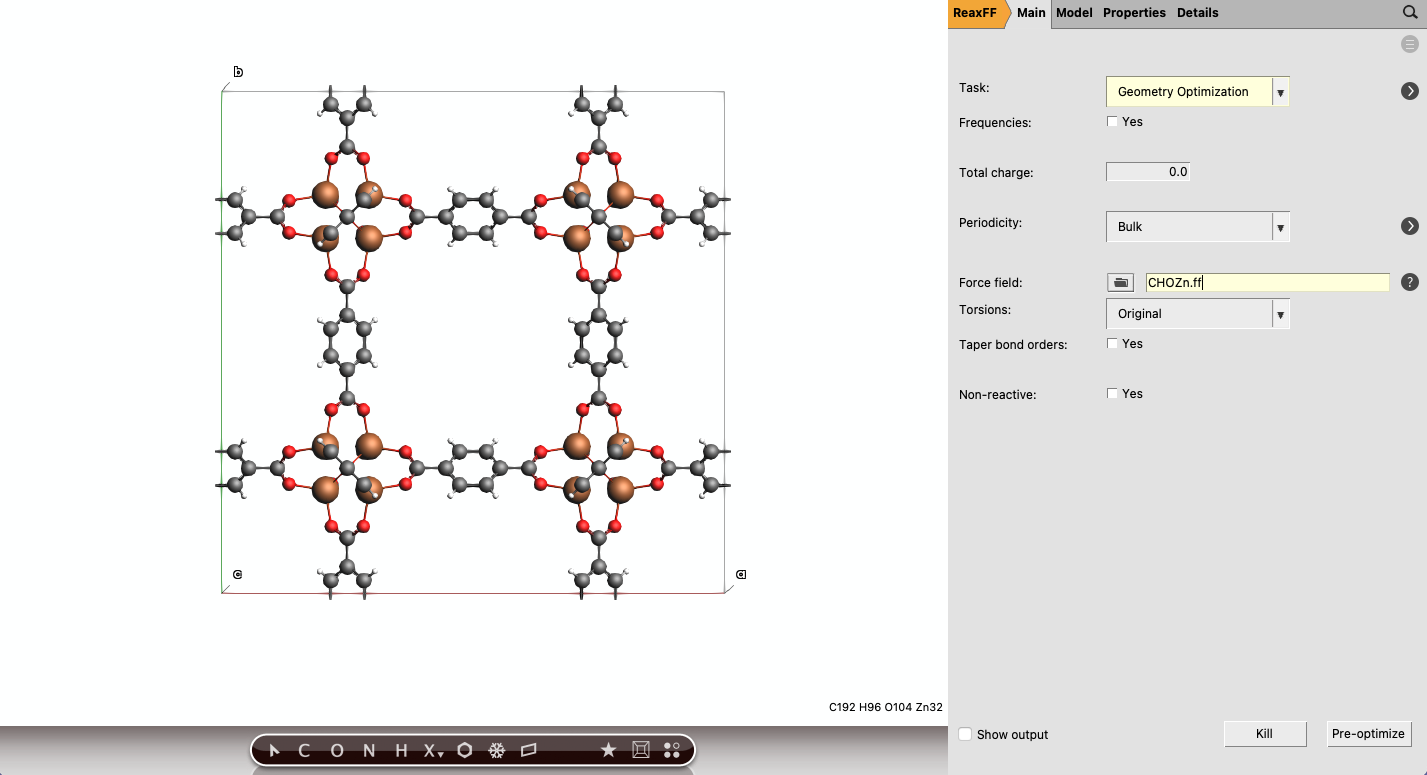
IRMOF-1
点开链接,复制所有数据,在 AMSinput Ctrl v粘贴(软件会提示“Use new lattice vector?”,点击Yes),即得到 IRMOF-1 结构。
优化结构:
如果压强不为0,则优化时,应在 Details → Geometry Optimization 窗口,勾选 Optimize Lattice,且在 Pressure 栏输入压强。
保存并运行作业。例如作业名为:IRMOF-1_ref_ReaxFF。
在指定温度、压强下的化学势计算
GCMC 模拟时,需要提供在指定的这个温度、压强下,体系的化学势。化学势 μ(T,P) 的通用定义如下:
\[\begin{split}\mu(T,P) = E_{CO2}+\Delta\mu(T,P_0)+RT\ln{\frac{P}{P_0}}\\ \text{其中 }\Delta\mu(T,P_0) = H(T,P_0)-H(0,P_0)-T\times \left[S(T,P_0)-S(0,P_0)\right]\end{split}\]
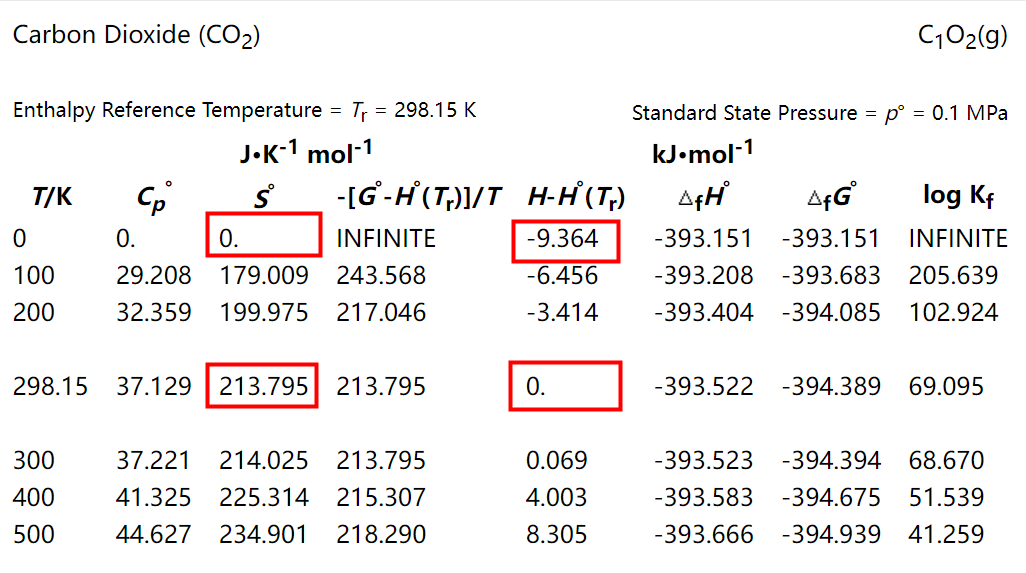
CO$_2$ 在不同温度下的 H 和 S 的值可以在 NIST 网站上直接查到。
298.15 K 时,不同压力下的 H 和 S 查询结果如下:
可以看到计算 Δμ(T,P$_0$) 所需的几个数据,T = 298.15K 时:
H(T,P$_0$) - H(0,P$_0$) = H(298.15K,P$_0$) - H(0K,P$_0$) 即表中 H-H$^。$(T$_r$) 这一列的两个数据,因此二者之差为:0.0 - (-9.364) kJ·mol$^{-1}$
S(T,P$_0$) - S(0,P$_0$) = H(298.15K,P$_0$) - H(0K,P$_0$) = 0.213795 - 0 kJ/(K·mol)
注意表中的数据是 213.795 J/(K·mol) 换算单位为 kJ/(K·mol),因此为 0.213795 kJ/(K·mol)
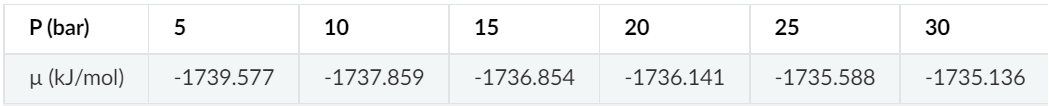
带入上文中 Δμ(T,P$_0$) 与 μ(T,P) 的计算公式中,压强 P = 30 bar 时:
\[\begin{split}\Delta\mu(298.15\text{ K},1.01\text{ bar}) = \left(0.0 - \left(-9.364\right)\right) - 298.15\times\left(0.213795 - 0.0\right) = -54.379\text{ kJ/mol} \\ \mu(298.15\text{ K},30\text{ bar}) = -1689.155 - 54.379 + 0.008314 \times 298.15 \times \ln{\frac{30.0}{1.01325}} = -1735.136\text{ kJ/mol}\end{split}\]
其中 E$_{CO2}$ 即前面优化 CO$_2$ 得到的能量 -1689.155kJ/mol,常数 R = 0.008314。类似地计算298.15K,压强为 5、10、15、20、25、30 bar 时,化学势为:
注:
- 上表中查阅到的 298K 以上的焓值和熵值,也可以通过 Shomate 方程计算得到,具体参考 NIST 网站(不过需要注意该网站无法算出 298K 以下温度的数据,而 0K 的数据,在我们上述计算中是需要用到的,因此仅供参考),这个网站有时候国内打不开,不过过一会儿又好了。
- 根据焓和熵的来源,以及您使用的精度和转换因子,您独立计算的数字可能略有不同。
巨正则系综蒙特卡洛模拟
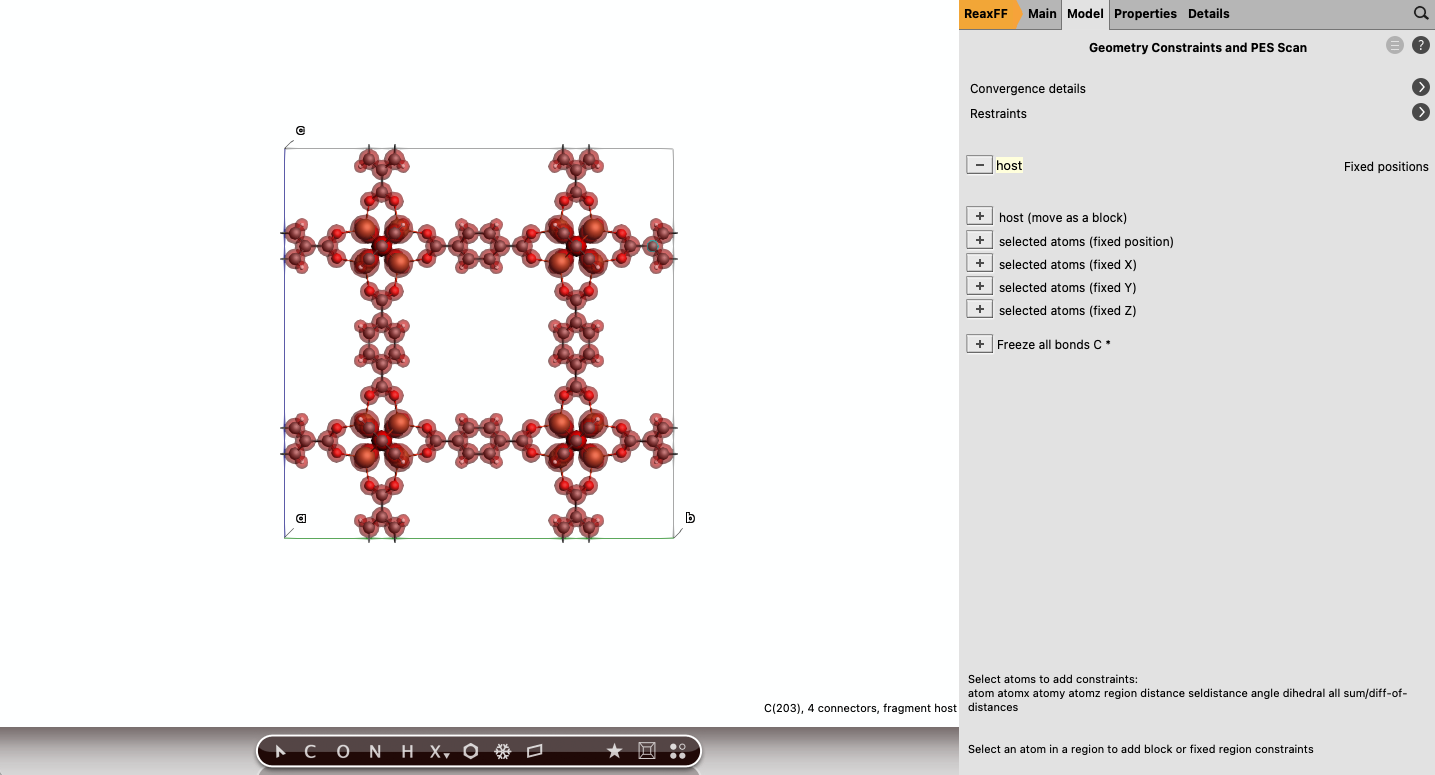
可以直接从前面优化 IRMOF-1 的窗口开始,重新打开该窗口时,会询问是否 update the coordinates,点击 Yes 更新为优化后的结构,Task 从 Geometry Optimization 改为 GCMC。后面的模拟,我们将冻结主体材料 IRMOF-1,将整个 IRMOF-1 创建为 Region,例如名为 host。然后 Model → Geometry Constraints and PES Scan,选中一个原子后,点击 (fixed position) 前面的 +:
然后设置 GCMC 相关的参数 Model → GCMC:
- Number of GCMC:100000
- Temperature:298.15 K
- Add molecules within:5.0
- Add molecules not closer than:1.2
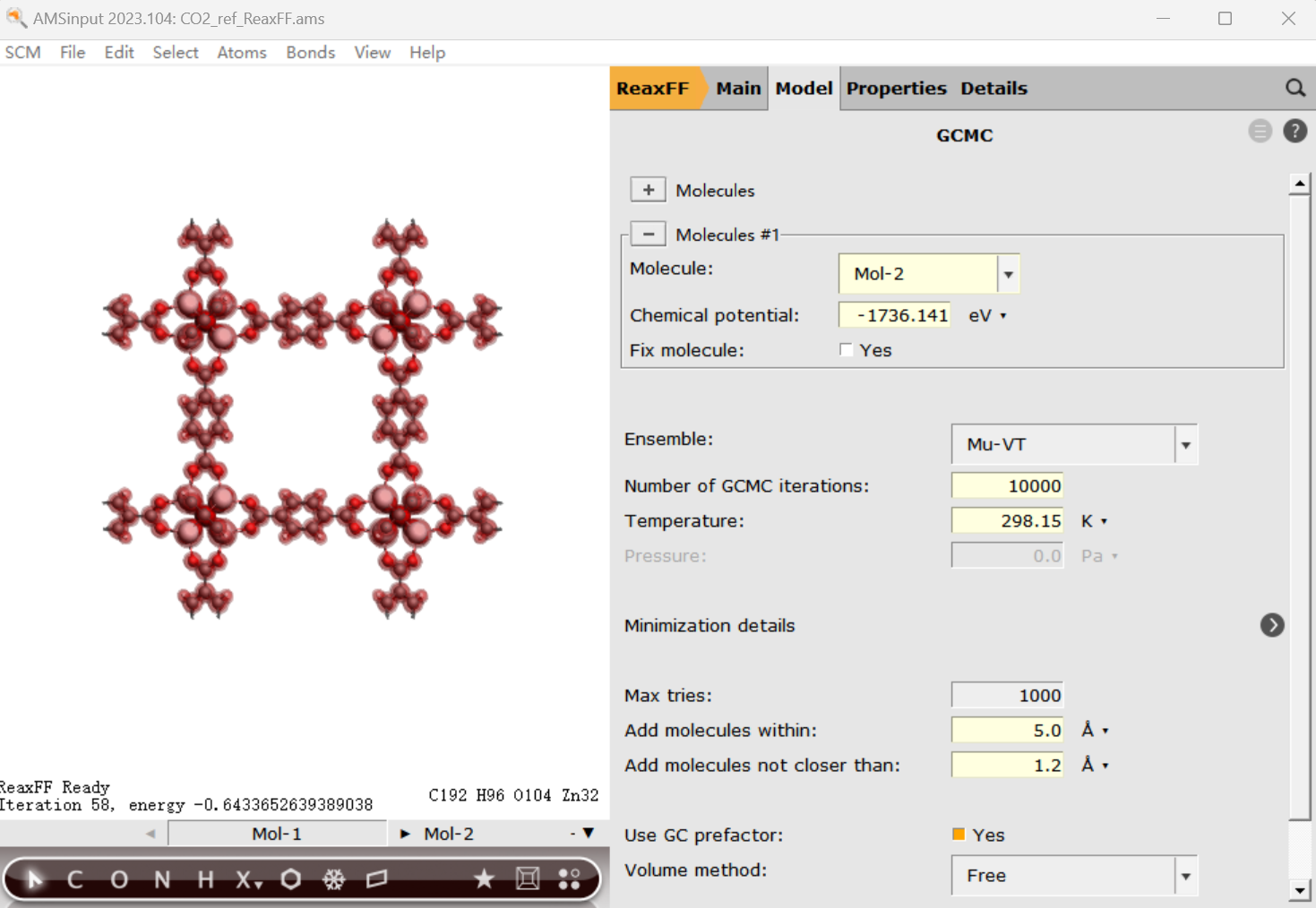
然后设置负载分子 CO$_2$ 相关参数,点击 Molecules 前的 +,新产生的 Molecule 选项选择 New Molecule,如此左边窗口就新增了一个窗口,底部mol-1、mol-2可以切换,其中 mol-1 应为 IRMOF-1,那么在 mol-2 中可以将之前优化得到的 CO$_2$ 粘贴进来。如此 Molecule 选项就变为了 Mol-2,如果我们关心的是 20 bar 下的情况,则Chemical potential 输入 该压强下的化学势 -1736.141 kJ/mol:
点击上图中 Minimization details 后面的 > 按钮,将 Convergence 选项改为 Basic,Maximum number of iterations 设置为 5000,如此使得 GCMC 较为容易收敛。这里如果最大迭代次数太小的话,得到的能量偏高,最终负载会被低估。而由于要优化的原子非常多(自由度非常大,尤其是插入了一定数目的客体分子后,自由度数量为6倍原子数,自由度越大,收敛越困难),因此需要较大的迭代步数才能收敛,而耗时也就越大,用户自己的体系,可以考虑开始设为1000步,如果收敛性不好,则可以提高到如本文一样5000步。
保存并运行作业,此计算可能需要几天时间。这里我们计算的是 20 bar,类似地,我们还需要进行不同压强下的计算,多个作业在同一个文件夹下更好,以便于后面使用 Python 对结果进行后处理。
用户下载并解压得到 analyze_gcmc.py 文件,将其放置于作业所在文件夹。AMSJobs窗口也进入作业所在文件夹,然后 Help → Command-line,打开命令行后,输入 sh 回车,输入命令运行该脚本:
amspython -u analyze_gcmc.py *.results/ams.rkf
如果有多个作业,可以在后面添加其他作业的ams.rkf文件。格式类似:amspython -u analyze_gcmc.py 1.results/ams.rkf 2.results/ams.rkf 3.results/ams.rkf ……
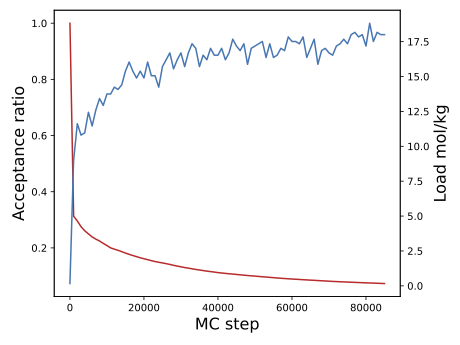
将得到类似如下接受率与负载曲线:
红色曲线为接受率(接受的 GCMC 步数与 GCMC步骤总数之比),蓝色曲线为负载情况。可以看到从 40000 步到 80000 步,基本上逐渐达到收敛,因为我们只计算了 10 万步,最终负载大约 17 mol/kg 左右。
总结
综上,在 20 bar、298.15K 下,我们计算得到的负载约为 17mol/kg,与 Environ Sci Pollut Res (2014) 21:5427–5449 报道的实验负载19 mol/kg 非常接近。
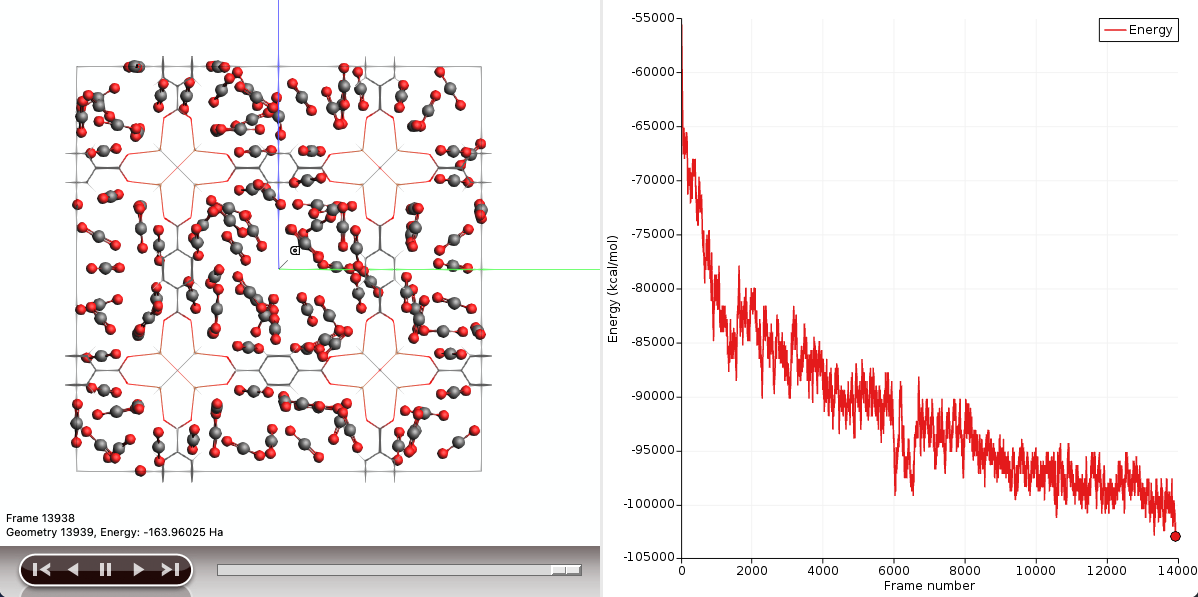
最终的负载情况优化得到的结构看起来如此:
不过可以看到实际上还没有完全收敛的样子,因此实际上用户可以尝试计算更多步数,例如 15~20 万步,或许可以得到更接近实验的结果。
最后,我们另外类似地计算5、10、15 bar 的吸附等温线,得到的4个值,绘制为曲线(如下图所示)。为了与实验结果比较,可以用最后 40000 个 GCMC 步的负载均值来作为极限载荷,误差条对应标准偏差。
我们可以得出结论:CHOZn.ff 很好地再现了 IRMOF-1 的实验等温线。
注意:与实验相比,其他体系的绝对等温线可能没有那么准确,不过如果力场得到了很好的优化(训练集中更好地覆盖的主体/客体的化学性质),至少相对等温线的预测会是有意义的(例如用于优化吸附剂)。对于生产质量等温线,建议 GCMC 步设置的足够大,并进行几个相同的独立作业,对几个作业的负载值进行平均,来作为该温度、压强下的负载。