这是本文档旧的修订版!
如何进行单重激发态的结构优化(非相对论)
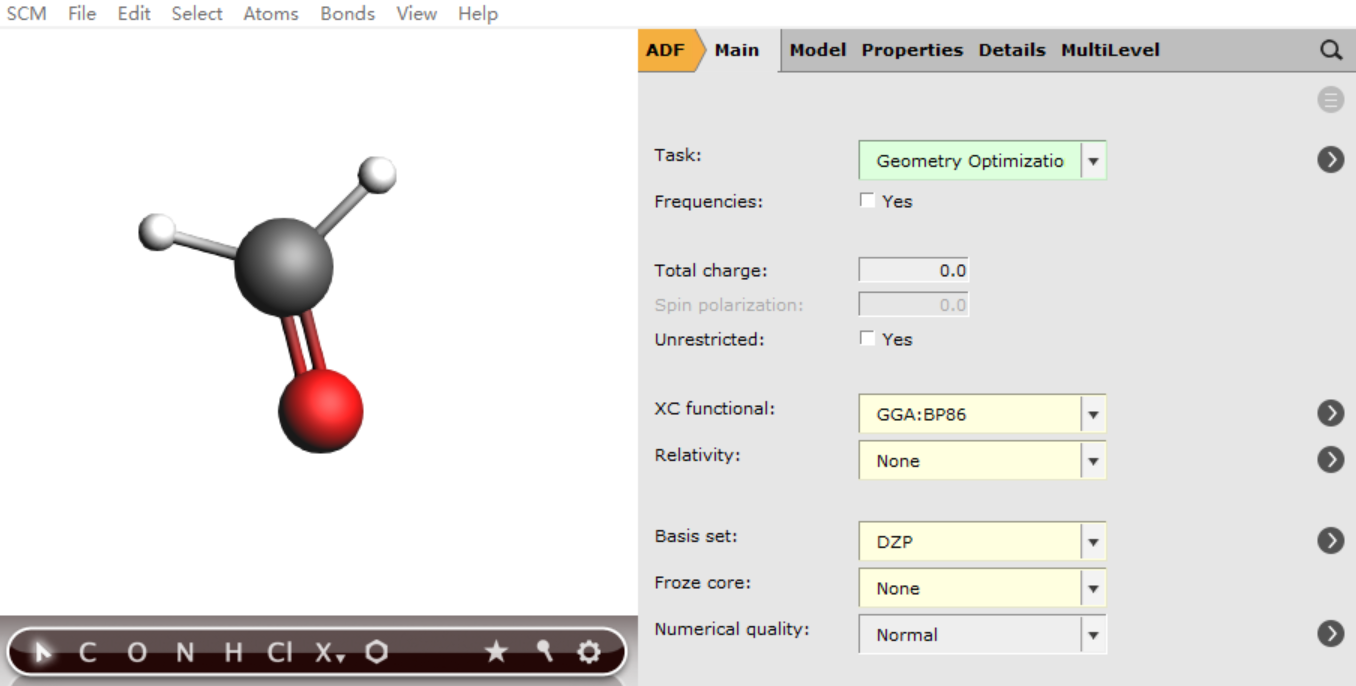
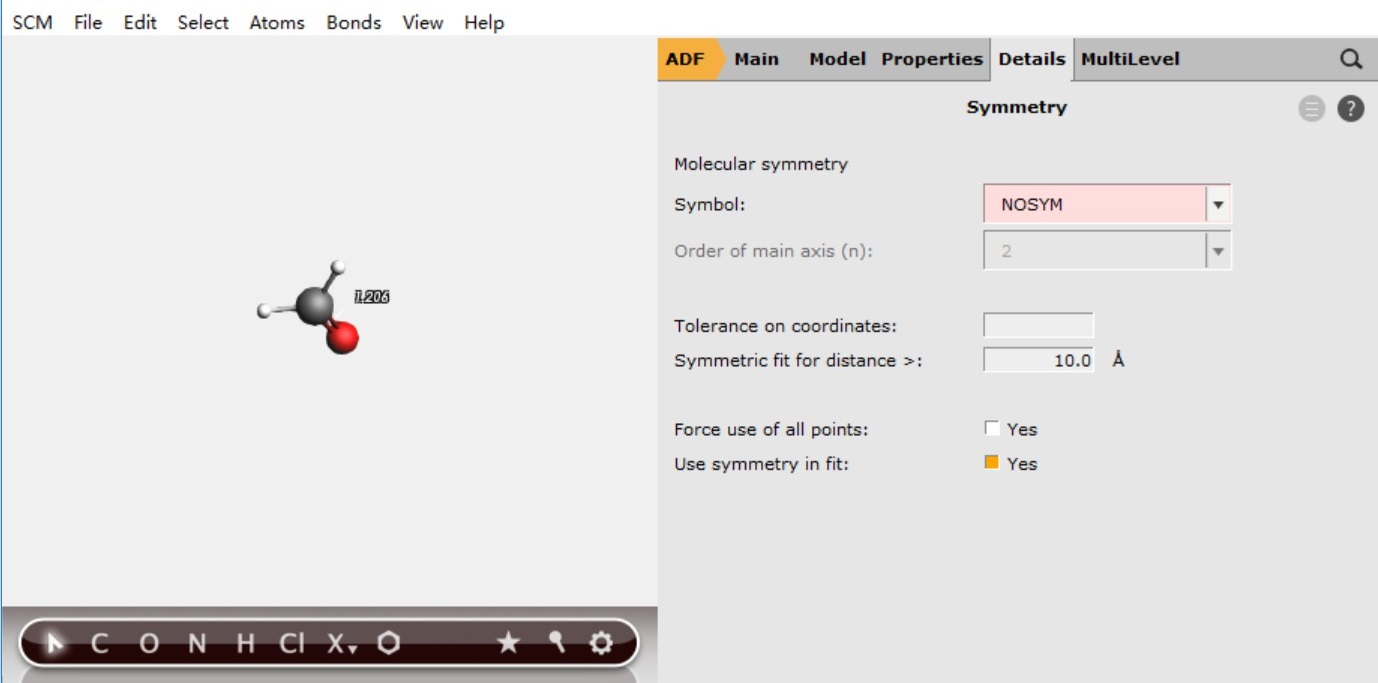
如果我们需要计算荧光或者磷光,激发态的结构优化就是必须要做的事情。如果计算吸收光谱就不需要这一步。一般而言,激发态的几何结构和基态的几何结构不一定具有相同的对称性。因此分子处于激发态的时候,其对称性我们是不清楚的。因此,对于激发态的优化,我们应该将分子的对称性取消掉,然后进行优化。优化之后,如果保持了原有的对称性结构,那就可以认为激发态对称性与基态一样(这种情况其实很少见);如果优化后,对称性消失了,或者降低了,那就只能认为激发态的对称性消失或降低了(大部分情况是如此);也有对称性变高的情况,但非常少。
ADF计算的精确度很高,因此能够让结构收敛到正确的对称性上面去。例如CO2分子,我们从一个对称性最低的结构,例如Cs群的结构去优化,最后收敛后,ADF也能得到D∞h的直线型结构。
由于我们取消了对称性,因此激发态的不可约表示就变成一个A了。最低激发态,也就变成1A了。同时最好也能细微移动其中一个对称原子,打破对称性,这样更加严谨。
本文的例子,没有考虑溶剂化,如果要考虑溶剂化,Model → Solvation选择溶剂化方法、溶剂即可,ADF新版支持溶剂化的激发态优化。
参数设置
我们如下设置激发态优化的参数(分子结构沿用上一步的结构)。激发态优化,不能使用Frozen Core,因此设置为None
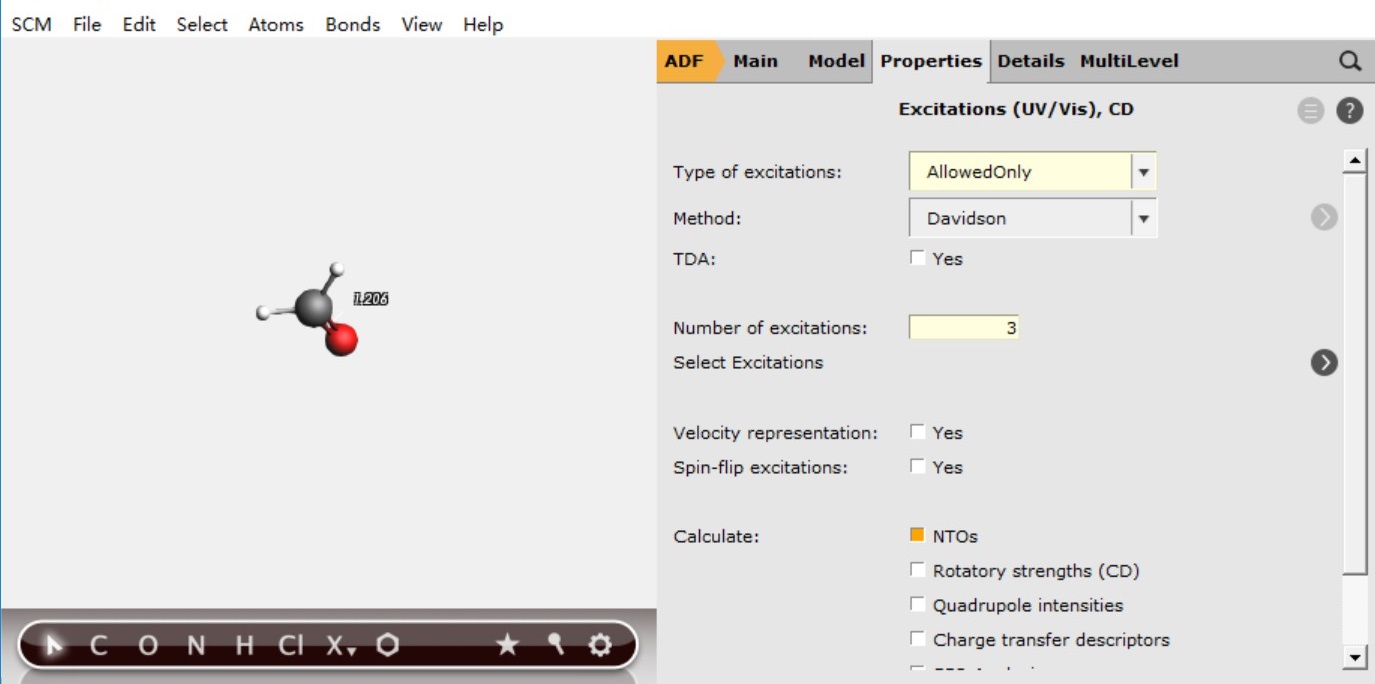
激发态的个数比需要计算的激发态序号大一点点即可:
注意,这里1 A表示优化第一个激发态,也就是S1;如果要优化Sn,那么这里就要填写n A。
结果分析
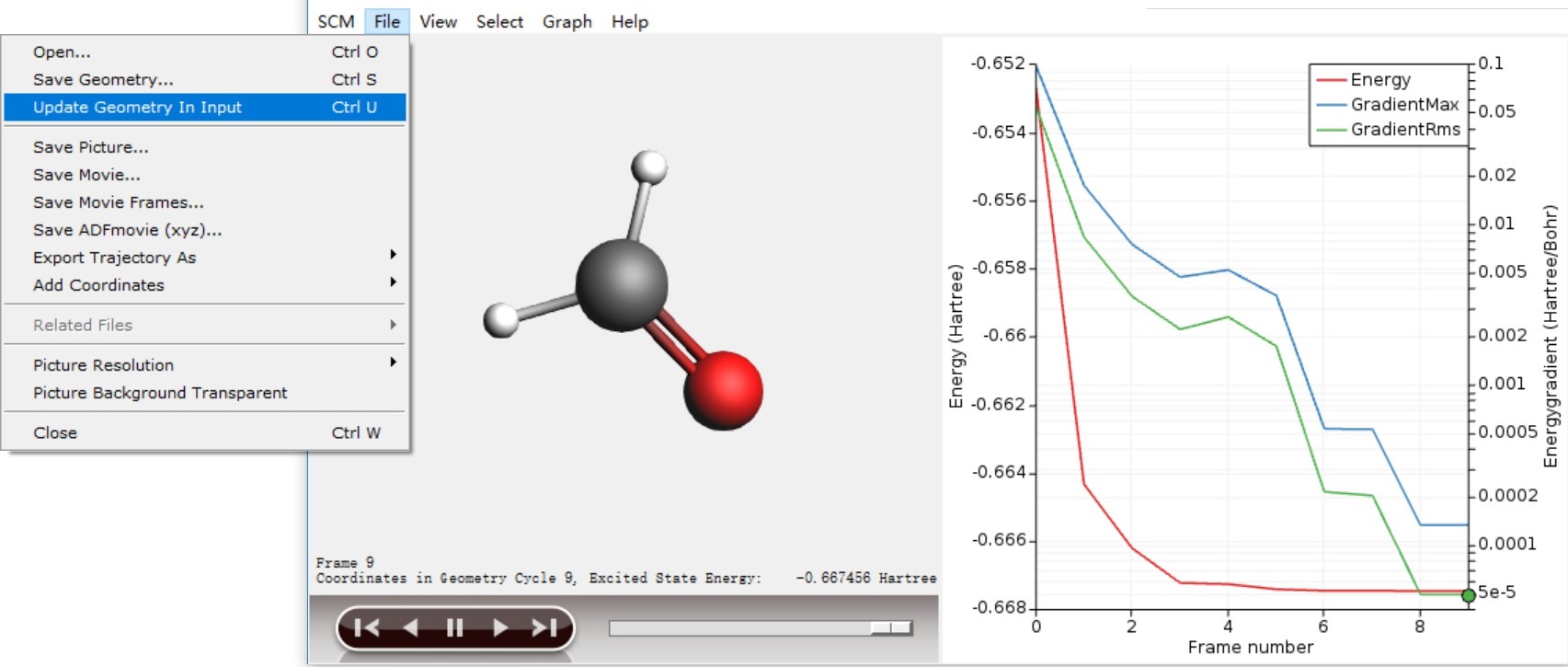
优化后的分子结构,可以从SCM LOGO > movie中播放的优化过程动画的最后一帧看到。导出该结构可以用如下方式:
也可以选择Save Geometry将其保存为xyz格式。
而优化结束之后的激发态,与前面一样,可以在SCM LOGO > Spectra中查看。可以看到新的S1态的激发能。这其实就对应着荧光的发射峰。组分等也可以同样查看到。
在新版中logfile会列出优化后得到的激发能:
<Dec01-2020> <19:56:03> New Excited State Energy: <Dec01-2020> <19:56:03> Total excited state energy: -0.66744908 Hartree <Dec01-2020> <19:56:03> NORMAL TERMINATION Job S1-geo has finished
但是,一定要注意:激发态优化过程中,是否存在势能面的交叉,也就是在SCM - Output,窗口底部输入“ All SINGLET-SINGLET excitation energies ”搜索激发能的变化。注意看我们计算的激发态的激发能,是否在过程中,出现与其他激发能差值接近0.1eV的情况,如果出现,则很可能出现了势能面交叉,应该另行考虑。
验证激发态优化结果:激发态频率计算,看是否存在虚频
参考:如何进行单重激发态的振动频率计算,注意激发态频率计算的泛函、基组、相对论,应该与激发态优化相同。
确认振动谱中没有虚频(负数的频率),则该激发态优化没有问题。