用python统计ReaxFF轨迹中的分子个数、键、氢键、环的数目变化
1,统计化学键
Python脚本右键另存下载并解压 该脚本只能分析AMS2023.101-2023.103之间的版本的ReaxFF输出轨迹文件*.results/ams.rkf。
运行方式:
- AMSjobs窗口中,进入该作业所在文件夹 → Help → Command-line → 打开命令行窗口输入sh回车(如果是Linux,本身就有命令行,直接打开命令行即可)
- 该脚本下载到某作业所在文件夹,与作业的*.resutls/同级
- 在命令行输入:amspython MoleculesTable-nPlots.py test.results/ams.rkf回车。稍后将产生分子数列表窗口,可以关闭:
- 然后会弹出化学键数窗口,也可以关闭:
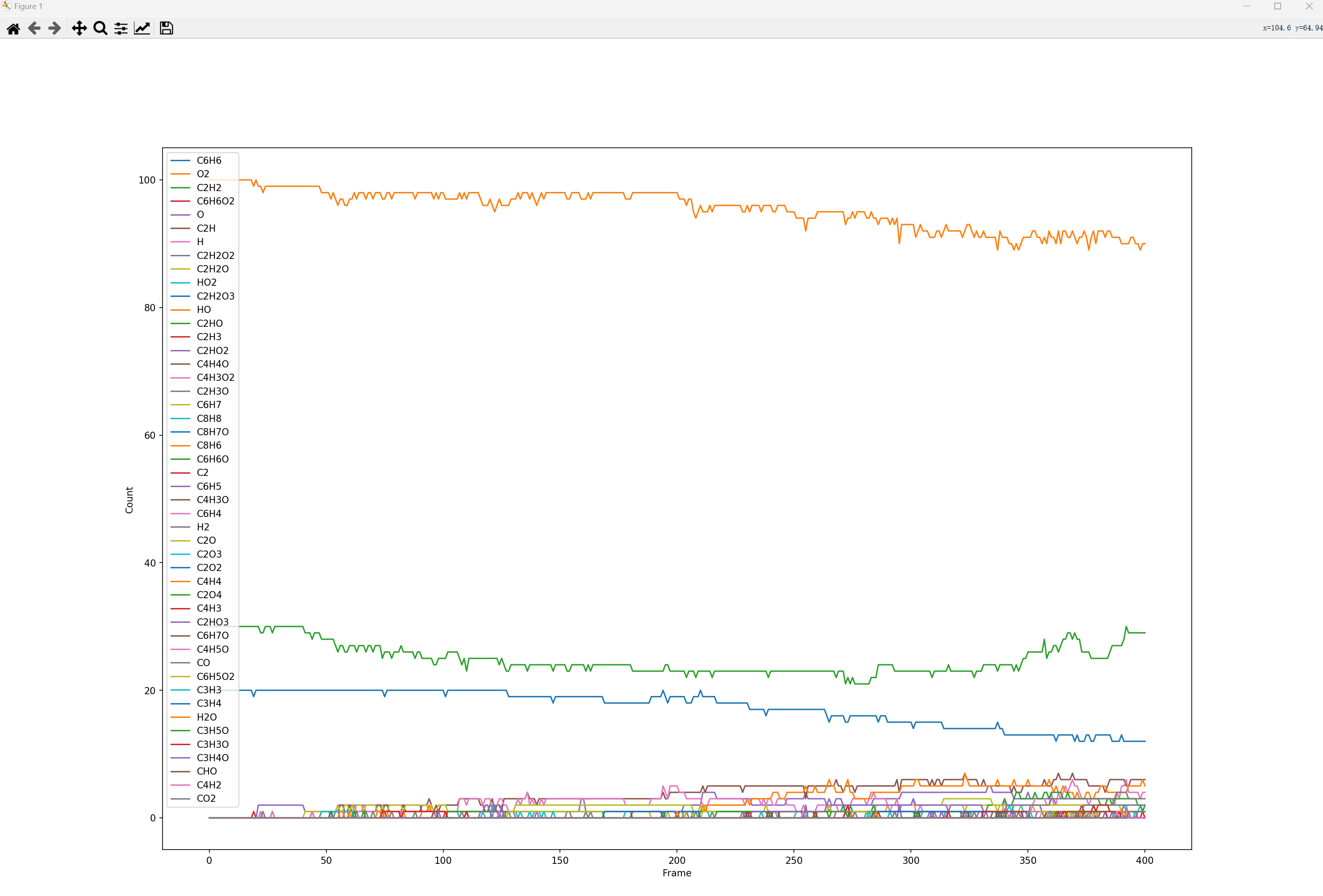
- 将生成:molecule_analysis.txt、molecule_analysis.pdf、bond_analysis.txt、bond_analysis.pdf四个文件。其中*.pdf文件是图,*.txt文件是曲线对应的数据,其中第一列为“帧”数,其他列为具体数据。
该脚本默认统计单、双、三键,如果需要统计芳香键,则修改脚本中:
round_type = 'integer' # round bond orders to nearest integer #round_type = 'half_integer' # round bond orders to nearest half integer
为
#round_type = 'integer' # round bond orders to nearest integer round_type = 'half_integer' # round bond orders to nearest half integer
注意
- 如果数据量很大,那么在一张图里面体现所有的分子数量、键的数量,就不太可能了。可以修改脚本中nplots = 的值更大,这样将会在多张图中显示这些曲线,这个值即图的数量。
- 最终会分别生成分子数量的*.txt文件与化学键数量的*.txt文件,用户也可以从这两个文件中的数据,自行作图。
2,增加环、氢键统计功能
2023.104以后的版本,在原先脚本功能的基础上增加了对环与氢键的统计。脚本右键另存下载并解压。在前面的版本基础上,增加了统计芳香环、氢键的功能。
ams.rkf文件可以是旧的,但是分析的时候,软件必须是最新的,因为python分析的时候,调用了一个库,2023.104及其之前的版本里面没有。
开关:
switch = {'Mol_analysis':True,'Bond_analysis':True,'Ring_analysis':True,'Hb_analysis':True}
Ture表示开关打开,False表示关闭。
氢键的种类需要自己定义一下:
donor_list = ['O'] acceptor_list = ['O'] d_hb_cutoff = 3.0 ang_hb_cutoff = 150.0
其中氢键表示为“D-H–A”,donor_list为“D”元素,acceptor_list为A元素,如果存在多种D、A可以在[]中列出,用逗号隔开,例如['O','N']。
运行过程中出现“No artists with labels found to put in legend. Note that artists whose label start with an underscore are ignored when legend() is called with no argument.”不用管它。